Restrikčná endonukleáza
Restrikčná endonukleáza (iné označenia: restrikčná nukleáza, restrikčný enzým, restriktáza [pozn 1]) je enzým, ktorý rozpoznáva DNA na základe určitej krátkej sekvencie a následne DNA štiepi. Z chemického hľadiska ide o hydrolýzu, restrikčné endonukleázy teda na štiepenie DNA na jednom mieste využívajú jednu molekulu vody a ako enzýmy sú zaraďované medzi hydrolázy. Niektoré restrikčné endonukleázy štiepia DNA na nešpecifických miestach, ktoré sú od rozpoznávanej sekvencie vzdialené (aj viac ako tisíc párov báz), iné štiepia DNA priamo na rozpoznávaných sekvenciách.[1][2]
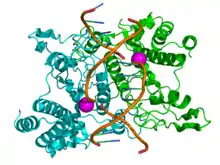
Restrikčné endonukleázy produkujú prokaryotické organizmy, baktérie aj archea a používajú ich na obranu proti vírusom. Endogénna DNA prokaryotických organizmov je totiž metylovaná a väčšina restrikčných endonukleáz ju preto nerozpoznáva - na rozdiel od DNA vírusov, ktorá metylovaná nie je. Takáto ochranná funkcia u prokaroyt je súčasťou restrikčno-modifikačného systému.[1] [2][3][4][5]
Nomenklatúra
Názov každej restrikčnej endonukleázy je skratkou pre organizmus, z ktorej bola izolovaná. Za touto skratkou nasleduje číslo, ktoré označuje, o koľkú restrikčnú endonukleázu izolovanú z daného organizmu ide. Napríklad názov restrikčnej endonukleázy EcoRI vychádza z baktérie E coli, písmeno R znamená bakteriálny kmeň RY13 a rímska číslica I znamená, že ide o prvú restrikčnú endonukleázu izolovanú z tohto bakteriálneho kmeňa. [1]
Rozpoznávaná sekvencia
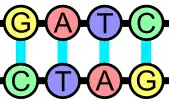
Sekvencia rozpoznávaná restrikčnými endonukleázami má obvykle dĺžku 4-8 párov báz. Počet nukleových báz, ktoré sú potrebné na rozpoznanie sekvencie DNA, určuje, aká je v ľubovoľnom genóme pravdepodobnosť, že dôjde k štiepeniu. Napr. špecifická sekvencia 4 párov báz má pravdepodobnosť výskytu 44, čiže každú 256. nukleovú bázu. ale sekvencia 8 párov báz má pravdepodobnosť výskytu 48, čiže každú 65 536. nukleovú bázu. Väčšina rozpoznávaných sekvencií je palindromických, poradie báz v jednom smere (napr. 5'-3') na jednom vlákne je teda zhodné s poradím báz na druhom vlákne v rovnakom smere (5'-3'). Restrikčné endonukleázy štiepia vždy každé vlákno DNA na rovnakom mieste v sekvencii daného vlákna. Ak je štiepenie symetrické, sú obe vlákna DNA rozštiepené v strede rozpoznávanej sekvencie a nové konce dvojzávitnice DNA sa označujú ako tzv. tupé konce. Tupé konce tvorí napr. restrikčná endonukleáza SmaI:
![]()
Naopak, ak restrikčná endonukleáza štiepi DNA asymetricky, vznikajú tzv. presahy a konce DNA sa označujú ako lepivé alebo kohézne. Niektoré restriktázy vytvárajú presah 5'-vláknami, iné 3' vláknami. Presah s 3' vláknami tvorí napr. restriktáza EcoRI:[1][2]
![]()
Restrikčné endonukleázy, ktoré rozpoznávajú rovnakú sekvenciu, ale DNA štiepia na rôznych miestach, sa nazývajú neoschizoméry. Rôzne restrikčné endonukleázy, ktoré navyše aj štiepia rovnakú sekvenciu, sa nazývajú izoschizoméry. [6]
Typy restrikčných endonukleáz
Na základe kofaktorov potrebných pre ich enzýmovú aktivitu a na základe rozpoznávanej sekvencie sa restriktázy rozdeľujú na tri typy, pričom typ II je dôležitý pre výskum v oblasti molekulárnej biológii, pretože sa využíva pre prácu s rekombinantnou DNA, konkrétne na molekulárne klonovanie. [1]
Typ I
Restrikčné endonukléazy typu I sú multifunkčné a viacpodjednotkové enzýmy. Skladajú sa z troch podjednotiek - HsdR, HsdM a HsdS. Podjednotka HsdS je zodpovedná za rozpoznanie špecifickej sekvencie DNA. Táto sekvencia sa v prípade typu I restrikčných endonukleáz skladá z dvoch miest na dvojzávitnici DNA - jedno je tvorené 3-4 nukleotidmi, druhé 4-5 nukleotidmi, pričom medzi týmito miestami je nešpecifická sekvencia približne 6 nukleotidov. Po rozpoznaní tejto sekvencie tieto restriktázy štiepia DNA na mieste vzdialenom minimálne 1000 párov báz od rozpoznanej sekvencie a táto hydrolytická aktivita je zabezpečená podjednotku HsdR. Podjednotka HsdM zabezpečuje týmto enzýmom schopnosť vykonávať metyláciu (metyl trasnferázová aktivita). Pre metyláciu potrebujú restrikčné endonukleázy typu I kofaktor S-adenozyl metionín, pre hydrolýzu DNA používajú ako kofaktory ATP a horečnaté ióny Mg2+.[7]
Typ II
Restrikčné endonukleázy typu II sú typicky jednoduché enzýmy, štiepia priamo sekvenciu DNA, ktorú rozpoznávajú a nemajú schopnosť metylovať DNA. Pre svoju aktivitu nepotrebujú ako kofaktor ani ATP, iba horečnaté ióny Mg2+. Ide o najpoužívanejšie restrikčné endunukleázy, ktoré sa používajú v molekulárnej biológii pre prácu s rekombinantnou DNA. Doposiaľ boli izolované tisícky restriktáz rozpoznávajúcich viac ako 100 špecifických sekvencii a s ich pribúdajúcim počtom a rozmanitosťou bolo zavedené podrobnejšie rozdelenie.[1]
Typické jednoduché restrikčné endonukleázy sú zaraďované do skupity restriktáz typu IIP. Typ IIB restriktáz sú multimery a DNA štiepia na dvoch miestach tak, že rozpoznanú sekvenciu vystrihnú z dvojzávitnice. Typ IIE potrebuje pre svoju aktivitu dve rovnaké sekvencie DNA, pričom štiepi iba jednu a druhú využíva ako alosterický regulátor. Typ IIF je podobný, štiepi však obe rozpoznávané sekvencie. Typ IIG sa najviac podobá typickým restriktázam typu II, pre svoju aktivitu však potrebujú S-adenozyl metionín. Typ IIM rozpoznáva iba špecifické sekvencie DNA, ktoré sú metylované. Restriktázy typu IIS štiepia DNA v presne definovanej vzdialenosti od rozpoznávanej sekvencie. TypT restrikčných endonukleáz funguje v podobe diméru, pričom každá podjednotka má jedno miesto pre rozpoznanie jedného vlákna DNA.[8]
Typ III
Restrikčné endonukleázy typu III rozpoznávajú dve nepalindromické sekvencie, ktoré sú navzájom orientované inverzne. DNA štiepia 20-30 párov báz za týmito sekvenciami. Ide o heteromérne proteíny skladajúce sa z dvoch podjednotiek - Res a Mod. Podjednotka Mod rozpoznáva sekvenciu DNA a má metylázovú aktivitu, ako kofaktor vyžaduje S-adenozyl metionín. Podjednotka Res má hydrolázovú aktivitu a štiepi DNA, ako kofaktory vyžaduje ATP a horečnaté ióny Mg2+. Samotné restriktázy typu III sú schopné rozpoznávať iba nemetylovanú DNA a metylujú iba jedno vlákno dvojzávitnice. [9]
Poznámky
- V praxi sa často vyskytujú tvary reštrikčná (endo)nukleáza a reštrikčný enzým. Správnosť tejto alternatívy je otázna: Podľa Veľkého slovníka cudzích slov (2000) je v tomto kontexte správne ako podstatné meno restrikcia alebo reštrikcia, ale ako prídavné meno len restrikčný (ak sa text slovníka berie doslova a nepredpokladá sa, že pri hesle reštrikčný sa pozabudlo na pridanie tohto biochemického významu). Slovenské kodifikačné príručky ani Slovník cudzích slov - akademický tento termín vôbec neuvádzajú. Slovo restriktáza sa však píše len so -s-.
Referencie
- NELSON, David L.; COX, Michael M.. Lehninger Principles of Biochemistry. 6. vyd. New York : W. H. Freeman and Company, 2013. ISBN 978-1-4641-0962-1. (anglicky)
- ALBERTS, Bruce; BRAY, Dennis; JOHNSON, Alexander, Julian Lewis, Martin Raff, Keith Roberts, Peter Walter Základy buněčné biologie. Redakcia Miranda Brownová, Eleanor Lawrenceová, Valerie Nealová, Anne Vinnicombeová; preklad Arnošt Kotyk, Bohumil Bouzek, Pavel Hozák; ilustrácie Nigel Orme. 2.. vyd. Ústí nad Labem : Espero Publishing, © 1998. ISBN 80-902906-2-0. (česky)
- Restriction endonucleases. CRC Critical Reviews in Biochemistry, 1976, s. 123–64. DOI: 10.3109/10409237609105456. PMID 795607.
- DNA modification and restriction. Annual Review of Biochemistry, 1969, s. 467–500. DOI: 10.1146/annurev.bi.38.070169.002343. PMID 4897066.
- Bacteriophage survival: multiple mechanisms for avoiding the deoxyribonucleic acid restriction systems of their hosts. Microbiological Reviews, 1983, s. 345–60. PMID 6314109.
- Isoschizomers [online]. 2015. Dostupné online.
- Type I restriction systems: sophisticated molecular machines (a legacy of Bertani and Weigle). Microbiology and Molecular Biology Reviews, 2000, s. 412–34. Dostupné online. DOI: 10.1128/MMBR.64.2.412-434.2000. PMID 10839821.
- Everything you wanted to know about type II restriction enzymes [online]. 2015. Dostupné online.
- Type III restriction enzymes need two inversely oriented recognition sites for DNA cleavage. Nature, Jan 1992, s. 467–9. DOI: 10.1038/355467a0. PMID 1734285.