Smithův–Watermanův algoritmus
Smithův-Watermanův algoritmus je jedním za základních algoritmů bioinformatiky. Provádí lokální zarovnání sekvencí (tedy takové, ve kterém jsou hledány nejpodobnější úseky všech možných délek mezi dvěma sekvencemi, oblasti na sekvencích vzdálené od těchto úseků jsou při hodnocení podobnosti zanedbány) dovolující mezery, nejčastěji jde o sekvence nukleotidů v nukleových kyselinách či sekvence aminokyselin v proteinech. Jde o algoritmus dynamického programování, sestavený Temple F. Smithem a Michaelem S. Watermanem v roce 1981.
Vlastnosti
Algoritmus vychází z algoritmu Needleman-Wunsch pro globální zarovnání – principiálně je velmi podobný, liší se hlavně položením všech negativních skóre rovno nule. Garantuje nalezení optimálního lokálního zarovnání vzhledem k parametrům (tedy skórovací matici a sankcím za vložení mezery (viz dále). Prostorová složitost je kde a jsou délky zarovnávaných sekvencí, časová složitost je kvadratická.
Princip
Vstupem algoritmu jsou 2 zarovnávané sekvence, skórovací matice a hodnota sankce za vložení mezery. Tradičně jde o celočíselné hodnoty. Algoritmus nejprve sestaví matici kde a jsou délky zarovnávaných sekvencí. Řádky matice přísluší jednotlivým znakům jedné sekvence, sloupce znakům druhé sekvence. První řádek a první sloupec nepřísluší žádnému znaku.
Postup:
= hodnota pole v matici na souřadnicích
= sankce za vložení mezery (0 nebo záporné číslo)
= hodnota za shodu/neshodu znaků příslušejících poli v matici na souřadnicích podle skórovací matice
- Do levého horního rohu matice zapíšeme hodnotu skóre .
- Do každého dalšího pole matice zapíšeme hodnotu (tedy nejvyšší skóre, jakého lze v tomto poli dosáhnout, nebo 0. Bude to buď skóre pole vlevo + sankce za vložení mezery (inzerce) nebo skóre pole nahoře + sankce za vložení mezery (delece) nebo skóre pole vlevo nahoře + hodnota za shodu/neshodu znaků na pozicích příslušných tomuto poli podle skórovací matice nebo, pokud jsou všechny tři předchozí hodnoty záporné, 0.) Zapamatujeme si, která z těchto možností byla oním maximem (tedy z kterého pole jsme se do tohoto dostali, při shodě si zapamatujeme všechny, pokud byla maximem 0, není třeba si nic pamatovat).
- Po zapsání všech takovýchto hodnot do všech polí matice začneme od pole s nejvyšším skóre (které jako poslední patří do nejlepšího lokálního zarovnání a zároveň je v něm zapsáno skóre tohoto zarovnání) a pozpátku postupně zjišťujeme, ze kterých polí jsme se do tohoto a každého z jeho předchůdců dostali, dokud nenarazíme na pole se skóre 0. Výsledná cesta v matici reprezentuje nejlepší zarovnání. Na rozdíl od globálního zarovnání může být výsledek kratší než obě srovnávané sekvence.
Příklad
Zarovnáváme nukleotidové sekvence ATCGAA a CATAC. Sankce za vložení mezery je . Skórovací matice vypadá takto:
(tedy hodnota za shodu/neshodu je pro všechny možnosti stejná. To nemusí platit vždy, především pro sekvence aminokyselin, kde se pravděpodobnost záměny mezi jednotlivými aminokyselinami liší)
Matice po dokončení algoritmu vypadá takto: (modré šipky naznačují předchůdce jednotlivých polí, červenými šipkami je vyznačena cesta optimálního zarovnání maticí)
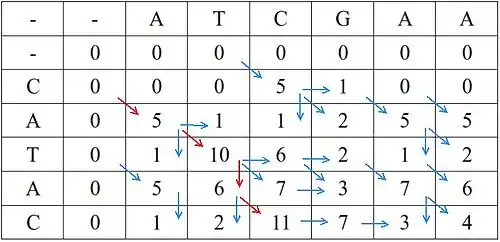
Výsledné zarovnání je tedy toto:
AT-CGAA || | CATAC
Skóre nejlepšího zarovnání je 11.
Použití
Algoritmus je vhodný na lokální zarovnání kratších sekvencí, mezi kterými jsou velké sekvenční či délkové rozdíly. Pro podobnější sekvence je častěji použito globální zarovnání algoritmem Needleman-Wunsch. Pro srovnávání dlouhých sekvencí na úrovni částí chromozomu jsou algoritmy dynamického programování nepoužitelné, kvůli příliš velké časové a prostorové náročnosti. V těchto případech se používají heuristické algoritmy jako BLAST, BLAT či PatternHunter, které nezaručují optimální zarovnání.
Je vidět, že matice obsahuje skóre všech možných lokálních zarovnání daných sekvencí s kladným skóre. Proto může být algoritmus použit k hledání různých zarovnání či pro srovnávání a hodnocení jiných (heuristických) algoritmů, které nemusí najít optimální řešení.